© Borgis - Postępy Nauk Medycznych 4/2015, s. 242-248
*Agnieszka Napiórkowska, Zofia Zwolska, Ewa Augustynowicz-Kopeć
Mutacje w genie pncA jako główny mechanizm oporności na pirazynamid wśród prątków gruźlicy**
Mutations in the pncA gene as a major mechanism of resistance to pyrazinamide among Mycobacterium tuberculosis
Zakład Mikrobiologii, Instytut Gruźlicy i Chorób Płuc, Warszawa
Kierownik Zakładu: prof. dr hab. n. med. Ewa Augustynowicz-Kopeć
Streszczenie
Wstęp. Postęp w oznaczaniu oporności na PZA dokonał się wraz z poznaniem genomu prątka oraz określeniem molekularnych mechanizmów oporności na lek, jakimi są mutacje w genie pncA. Gen pncA jest odpowiedzialny za prawidłową syntezę pirazynamidazy (PZaza) przekształcającej lek w jego aktywną formę. Mutacje w genie pncA występują na całej jego długości (561pz) oraz w regionie promotorowym. Mają one różnorodny charakter, najczęściej typu missense (skutkujące substytucją aminokwasu), ponadto insercje, delecje oraz mutacje typu nonsense.
Cel pracy. Celem pracy była identyfikacja mutacji w genie pncA występujących wśród prątków gruźlicy opornych na pirazynamid.
Materiał i metody. Analizie poddano 195 szczepów M. tuberculosis opornych na pirazynamid, które zostały wyizolowanie od polskich chorych w latach 2000-2011. Mutacje wykrywano, analizując sekwencje genu pncA i jego regionu promotorowego (700pz) w porównaniu z sekwencją „dzikiego” genu pncA szczepu M. tuberculosis H37Rv.
Wyniki. Analiza sekwencyjna genu pncA oraz regionu promotorowego wykazała, że 174 (89,2%) szczepy oporne na PZA posiadały mutacje w genie odpowiedzialnym za oporność na ten lek. Wykryto 57 różnych mutacji, które rozmieszczone były wzdłuż całego genu, ponad 60% szczepów wykazywało mutacje w części 3’ genu, między kodonem 86 a 187. W pracy zidentyfikowano 25 nowych mutacji, które do tej pory nie zostały zarejestrowane w bazie TB Drug Resistance Mutation Database.
Wnioski. Identyfikacja mutacji w genie pncA daje możliwość uzyskania w krótkim czasie informacji o PZA-oporności, co skutkuje szybkim włączeniem prawidłowego leczenia chorego na gruźlicę. Przedstawiona w pracy analiza może być podstawą do opracowania wiarygodnych i szybkich testów molekularnych do wykrywania PZA-oporności.
Summary
Introduction. Progress in the determination of resistance to PZA was made while learning the genome of the mycobacterium and molecular mechanisms of the resistance to the drug, which are mutations in the pncA gene. The pncA is responsible for the proper synthesis of pyrazinamidase (PZase) which converts the drug into its active form. The pncA mutations are highly diverse and scattered along the gene (561 bp) as well as the promoter region. They have diverse character, mostly missense causing amino acid substitutions, in some cases nucleotide insertions or deletions and nonsense mutations.
Aim. The aim of the study was to identify mutations in the pncA gene among PZA-resistant M. tuberculosis strains.
Material and methods. This study included 195 PZA-resistant M. tuberculosis strains isolated from Polish patients in the years 2000-2011. Mutations in the pncA gene were detected by comparing DNA sequences with the corresponding sequence of a wild-type pncA gene of M. tuberculosis H37Rv.
Results. The sequence analysis of the pncA gene revealed that 174 (89.2%) strains had mutation. 57 different mutations were detected and they were dispersed along the pncA gene but more than 60% of the strains showed mutations in the 3’ part of the gene (between 86 and 186 codon). Among 57 different mutations 25 not previously reported in TB Drug Resistance Mutation Database were found.
Conclusions. Analysis of the pncA gene provides rapid information regarding PZA susceptibility in M. tuberculosis and may therefore contribute to early optimization of treatment. Analysis of mutations in the pncA gene in M. tuberculosis strains may contribute to the development of reliable and rapid molecular tests for detection of PZA resistance.

Wstęp
Pirazynamid (PZA) należy do grupy głównych leków przeciwprątkowych, który razem z izoniazydem, rifampicyną i etambutolem stosowany jest w rekomendowanym schemacie leczenia. Pomimo że zajmuje bardzo ważne miejsce w leczeniu gruźlicy, WHO nie rekomenduje oznaczenia oporności na ten lek, co wynika głównie z trudności w wykonaniu testu lekooporności na PZA. Postęp w oznaczaniu PZA-oporności dokonał się wraz z poznaniem genomu prątka oraz molekularnych mechanizmów oporności na lek (1, 2).
Podstawowym mechanizmem oporności prątków gruźlicy na pirazynamid są mutacje w genie pncA (561 pz). Gen pncA odpowiedzialny jest za prawidłową syntezę pirazynamidazy (PZasa) przekształcającej lek w jego aktywną formę (2-4). Wśród szczepów PZA-opornych enzym ten nie wykazuje swojej aktywności lub jego aktywność jest niższa niż u szczepów wrażliwych (2).
Mutacje odpowiedzialne za oporność na PZA występują wzdłuż całego genu pncA i obszaru promotorowego oraz mają różnorodny charakter. Najczęściej są to mutacje typu missense skutkujące substytucją aminokwasu, ale również insercje, delecje oraz mutacje typu nonsense (1, 5). Liczne badania pokazują, że istnieje kilka obszarów w genie pncA, które w znaczący sposób mogą wpływać na aktywność pirazynamidazy. Mutacje występujące w pierwszym kodonie (Met1) genu pncA zakłócają proces inicjacji translacji (6, 7). Mutacje w kodonie Asp8, który jest częścią triady katalitycznej Cys138-Asp8-Lys96, wpływają na zaburzenie jej prawidłowego funkcjonowania, a co za tym idzie utrudniają proces wiązania substratu podczas katalizy, podobnie w przypadku mutacji w kodonach Lys96 oraz Cys138. Ostatnie badania nad strukturą krystaliczną białka PncA wyizolowanego od M. tuberculosis wykazały, że większość obserwowanych mutacji wpływa na zmniejszenie stabilności struktury białka, co w konsekwencji powoduje obniżoną aktywność enzymu. Mutacje dotyczące aminokwasów występujących zarówno w aktywnym centrum katalitycznym, jak i w miejscu wiązania jonów żelaza czy miejscu wiązania substratu są mutacjami bezpośrednio zaangażowanymi w oporność na pirazynamid. Mutacje w kodonach Asp49, His51, His57 oraz His71 skutkują zmianą w miejscu wiązania jonu żelaza. Mutacje w kodonach Asp8, Val9, Thr47, Gly132, Ala134, Cys138 oraz Val139 wpływają na centrum aktywne enzymu, w wyniku czego powodują utratę jego aktywności. Zmiany w kodonach Gln10, Asp12, Ser104 oraz Thr142 zakłócają tworzenie wiązań wodorowych między atomami łańcucha głównego i łańcuchów bocznych białka. Wszystkie z wymienionych mutacji w istotny sposób wpływają na aktywność enzymatyczną białka PncA (8).
Cel pracy
Celem pracy była identyfikacja mutacji w genie pncA występujących wśród prątków gruźlicy opornych na pirazynamid.
Materiał i metody
Materiał do badań stanowiło 195 szczepów M. tuberculosis opornych na pirazynamid, które zostały wyizolowanie w latach 2000-2011 od chorych pochodzących z różnych rejonów Polski.
Mutacje w genie pncA wykrywano, sekwencjonując gen wraz z jego regionem promotorowym (fragment 700 pz), który otrzymywano w wyniku amplifikacji techniką PCR przy użyciu pary starterów pncAf (5’-GCTGGTCATGTTCGCGATCG-3’) i pncAr (5’-GCTTGCGGCGAGCGCTCCA-3’) (9). Otrzymane produkty PCR oczyszczano z zastosowaniem zestawu ExoSAP-IT (GE Healthcare, United States), a następnie sekwencjonowano przy użyciu automatycznego analizatora DNA typu ABI PRISM 310 (Applied Biosystems, CA, USA) w Zakładzie Sekwencjonowania DNA firmy Genomed. Do sekwencjonowania używano tych samych primerów, które stosowano w amplifikacji genu pncA metodą PCR. Analizę otrzymanych sekwencji nukleotydowych prowadzono przy użyciu programu Chromas 1.45 oraz programu Mutation Surveyor ver. 3.30 (SoftGenetics LLC, USA). Sekwencje analizowano w odniesieniu do sekwencji „dzikiego” genu pncA szczepu M. tuberculosis H37Rv dostępnej w bazie GenBank (kod dostępu: NC_018078).
Wyniki
Wśród analizowanej puli 195 szczepów M. tuberculosis opornych na PZA, u 174 (89,2%) stwierdzono mutacje w genie pncA. Większość analizowanych szczepów posiadało mutację w części 3’ genu pncA, tj. między kodonem 86 a 186. Mutacje w kodonach 86-131 stwierdzono u 36 szczepów (18,5%), w kodonach 132-142 u 21 (10,8%), w kodonach 142-186 u 53 szczepów (27,2%). Mutacje w genie pncA stwierdzono również w kodonach: 1-4 (3 szczepy – 1,5%), 5-12 (17 szczepów – 8,7%), 13-68 (20 szczepów – 10,3%), 69-85 (14 szczepów – 7,2%) oraz w regionie promotorowym (10 szczepów – 5,1%) (ryc. 1).
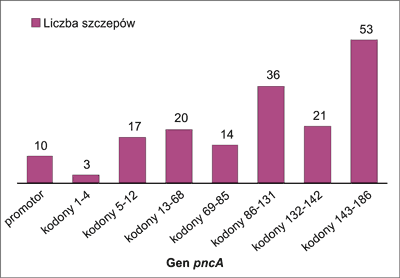
Ryc. 1. Miejsce występowania mutacji w genie pncA wśród prątków gruźlicy opornych na PZA.
Powyżej zamieściliśmy fragment artykułu, do którego możesz uzyskać pełny dostęp.
Mam kod dostępu
- Aby uzyskać płatny dostęp do pełnej treści powyższego artykułu albo wszystkich artykułów (w zależności od wybranej opcji), należy wprowadzić kod.
- Wprowadzając kod, akceptują Państwo treść Regulaminu oraz potwierdzają zapoznanie się z nim.
- Aby kupić kod proszę skorzystać z jednej z poniższych opcji.
Opcja #1
29 zł
Wybieram
- dostęp do tego artykułu
- dostęp na 7 dni
uzyskany kod musi być wprowadzony na stronie artykułu, do którego został wykupiony
Opcja #2
69 zł
Wybieram
- dostęp do tego i pozostałych ponad 7000 artykułów
- dostęp na 30 dni
- najpopularniejsza opcja
Opcja #3
129 zł
Wybieram
- dostęp do tego i pozostałych ponad 7000 artykułów
- dostęp na 90 dni
- oszczędzasz 78 zł
Piśmiennictwo
1. Napiórkowska A: Charakterystyka fenotypowa i molekularna szczepów Mycobacterium tuberculosis opornych na pirazynamid (PZA) wyizolowanych od chorych na gruźlicę w Polsce, w latach 2000-2011. Rozprawa doktorska, IGiChP, Warszawa 2014.
2. Zhang Y, Mitchison D: The curious characteristics of pyrazinamide: a review. Int J Tuberc Lung Dis 2003; 7(1): 6-21.
3. Campbell PJ, Morlock GP, Sikes RD et al.: Molecular detection of mutations associated with first- and second-line drug resistance compared with conventional drug susceptibility testing of Mycobacterium tuberculosis. Antimicrob Agents Chemother 2011; 55(5): 2032-2041.
4. Hou L, Osei-Hyiaman D, Zhang Z et al.: Molecular characterization of pncA gene mutations in Mycobacterium tuberculosis clinical isolates from China. Epidemiol Infect 2000; 124(2): 227-232.
5. Marttila HJ, Marjamäki M, Vyshnevskaya E et al.: pncA mutations in pyrazinamide-resistant Mycobacterium tuberculosis isolates from northwestern Russia. Antimicrob Agents Chemother 1999; 43(7): 1764-1766.
6. Lee KW, Lee JM, Jung KS: Characterization of pncA mutations of pyrazinamide-resistant Mycobacterium tuberculosis in Korea. J Korean Med Sci 2001; 16(5): 537-543.
7. Portugal I, Barreiro L, Moniz-Pereira J et al.: pncA mutations in pyrazinamide-resistant Mycobacterium tuberculosis isolates in Portugal. Antimicrob Agents Chemother 2004; 48(7): 2736-2738.
8. Muthaiah M, Jagadeesan S, Ayalusamy N et al.: Molecular Epidemiological Study of Pyrazinamide-Resistance in Clinical Isolates of Mycobacterium tuberculosis from South India. Int J Mol Sci 2010; 11(7): 2670-2680.
9. Somoskovi A, Dormandy J, Parsons LM et al.: Sequencing of the pncA gene in members of the Mycobacterium tuberculosis complex has important diagnostic applications: Identification of a species-specific pncA mutation in „Mycobacterium canettii” and the reliable and rapid predictor of pyrazinamide resistance. J Clin Microbiol 2007; 45(2): 595-599.
10. Napiórkowska A, Rüsch-Gerdes S, Hillemann D et al.: Characterisation of pyrazinamide-resistant Mycobacterium tuberculosis strains isolated in Poland and Germany. International Journal of Tuberculosis and Lung Disease 2014; 18(4): 454-460.
11. Sreevatsan S, Pan X, Zhang Y et al.: Mutations associated with pyrazinamide resistance in pncA of Mycobacterium tuberculosis complex organisms. Antimicrob Agents Chemother 1997; 41(3): 636-640.
12. Rodrigues VF, Telles MA, Ribeiro MO et al.: Characterization of pncA mutations in pyrazinamide-resistant Mycobacterium tuberculosis in Brazil. Antimicrob Agents Chemother 2005; 49(1): 444-446.
13. Clemente WT, Soares Lima SS, Palaci M et al.: Phenotypic and genotypic characterization of drug-resistant Mycobacterium tuberculosis strains. Diagn Microbiol Infect Dis 2008; 62(2): 199-204.
14. Pandey S, Newton S, Upton A et al.: Characterisation of pncA mutations in clinical Mycobacterium tuberculosis isolates in New Zealand. Pathology 2009; 41(6): 582-584.
15. Scorpio A, Lindholm-Levy P, Heifets L et al.: Characterization of pncA mutations in pyrazinamide-resistant Mycobacterium tuberculosis. Antimicrob Agents Chemother 1997; 41(3): 540-543.
16. Lemaitre N, Sougakoff W, Truffot-Pernot C et al.: Characterization of new mutations in pyrazinamide-resistant strains of Mycobacterium tuberculosis and identification of conserved regions important for the catalytic activity of the pyrazinamidase PncA. Antimicrob Agents Chemother 1999; 43(7): 1761-1763.
17. Barco P, Cardoso RF, Hirata RD et al.: pncA mutations in pyrazinamide-resistant Mycobacterium tuberculosis clinical isolates from the southeast region of Brazil. J Antimicrob Chemother 2006; 58(5): 930-935.
18. Stoffels K, Mathys V, Fauville-Dufaux M et al.: Systematic analysis of pyrazinamide-resistant spontaneous mutants and clinical isolates of Mycobacterium tuberculosis. Antimicrob Agents Chemother 2012; 56(10): 5186-5193.
19. Cheng SJ, Thibert L, Sanchez T et al.: pncA mutations as a major mechanism of pyrazinamide resistance in Mycobacterium tuberculosis: spread of a monoresistant strain in Quebec, Canada. Antimicrob Agents Chemother 2000; 44(3): 528-532.
20. Escalante P, Ramaswamy S, Sanabria H et al.: Genotypic characterization of drug-resistant Mycobacterium tuberculosis isolates from Peru. Tuber Lung Dis 1998; 79(2): 111-118.
21. Park SK, Lee JY, Chang CL et al.: pncA mutations in clinical Mycobacterium tuberculosis isolates from Korea. BMC Infect Dis 2001; 1: 4.
22. Doustdar F, Khosravi AD, Farnia P: Mycobacterium tuberculosis genotypic diversity in pyrazinamide-resistant isolates of Iran. Microb Drug Resist 2009; 15(4): 251-256.
23. Jurèen P, Werngren J, Toro JC et al.: Pyrazinamide resistance and pncA gene mutations in Mycobacterium tuberculosis. Antimicrob Agents Chemother 2008; 52(5): 1852-1854.
24. Yüksel P, Tansel O: Characterization of pncA mutations of pyrazinamide-resistant Mycobacterium tuberculosis in Turkey. New Microbiol 2009; 32(2): 153-158.
25. Mestdagh M, Fonteyne PA, Realini L et al.: Relationship between pyrazinamide resistance, loss of pyrazinamidase activity, and mutations in the pncA locus in multidrug-resistant clinical isolates of Mycobacterium tuberculosis. Antimicrob Agents Chemother 1999; 43(9): 2317-2319.
26. Chan RC, Hui M, Chan EW et al.: Genetic and phenotypic characterization of drug-resistant Mycobacterium tuberculosis isolates in Hong Kong. J Antimicrob Chemother 2007; 59(5): 866-873.
27. Sheen P, Mèndez M, Gilman RH et al.: Sputum PCR-single-strand conformational polymorphism test for same-day detection of pyrazinamide resistance in tuberculosis patients. J Clin Microbiol 2009; 47(9): 2937-2943.
28. Morlock GP, Crawford JT, Butler WR et al.: Phenotypic characterization of pncA mutants of Mycobacterium tuberculosis. Antimicrob Agents Chemother 2000; 44(9): 2291-2295.
29. Sandgren A, Strong M, Muthukrishnan P et al.: Tuberculosis drug resistance mutation database. PLoS Med 2009; 6(2): e2.
30. Zhang Y, Scorpio A, Nikaido H et al.: Role of acid pH and deficient efflux of pyrazinoic acid in unique susceptibility of Mycobacterium tuberculosis to pyrazinamide. J Bacteriol 1999; 181(7): 2044-2049.
31. Shi W, Zhang X, Jiang X et al.: Pyrazinamide inhibits trans-translation in Mycobacterium tuberculosis. Science 2011; 333(6049): 1630-1632.
32. Alexander DC, Ma JH, Guthrie JL et al.: Gene sequencing for routine verification of pyrazinamide resistance in Mycobacterium tuberculosis: a role for pncA but not rpsA. J Clin Microb 2012; 50(11): 3726-3728.
33. Simons SO, Mulder A, van Ingen J et al.: Role of rpsA gene sequencing in diagnosis of pyrazinamide resistance. J Clin Microb 2013; 51(1): 382.
34. Simons SO, van Ingen J, van der Laan T et al.: Validation of pncA gene sequencing in combination with the Mycobacterial Growth Indicator Tube method to test susceptibility of Mycobacterium tuberculosis to pyrazinamide. J Clin Microbiol 2012; 50(2): 428-434.
35. Alexander DC, Ma JH, Guthrie JL et al.: Reply to „Role of rpsA gene sequencing in diagnosis of pyrazinamide resistance”. J Clin Microb 2013; 51(1): 383.

