© Borgis - Nowa Stomatologia 3/2001, s. 15-18
Izabela Strużycka1, Anna Skoczyńska2, Katarzyna Rucińska1
Zastosowanie metody PCR do identyfikacji paciorkowców z grupy mutans
Identification of mutans streptococci by PCR method
1 z Zakładu Stomatologii Zachowawczej Instytutu Stomatologii Akademii Medycznej w Warszawie
Kierownik Zakładu: prof. dr hab. n. med. Maria Wierzbicka
2 z Centralnego Laboratorium Surowic i Szczepionek w Warszawie
Kierownik Laboratorium: prof. dr hab. med. Waleria Hryniewicz

Do grupy paciorkowców próchnicotwórczych mutans należy siedem gatunków (1, 2). Sposród tych gatunków Streptococcus mutans i Streptococcus sobrinus są najczęściej izolowane z jamy ustnej u ludzi i uważane są za najważniejsze bakteryjne czynniki etiologiczne próchnicy zębów u ludzi. Wyniki badań epidemiologicznych sugerują, że obecność S. sobrinus jest w większym stopniu związana z wysoką aktywnością próchnicy niż S. mutans (3, 4). W związku z tym wydaje się, że możliwość wykrywania tych dwóch mikroorganizmów może być interesująca z punktu widzenia zapobiegania i prognozowania próchnicy.
Identyfikacja drobnoustrojów z rodzaju Streptococcus do gatunku klasycznymi metodami jest złożona i czasochłonna. Z tego powodu opracowano komercyjne zestawy do szybkiej identyfikacji tych drobnoustrojów (5, 6). Równolegle z rozwojem szybkich metod diagnostycznych, wykorzystujących fenotypowe właściwości drobnoustrojów, nastąpił dynamiczny rozwój metod biologii molekularnej i co za tym idzie próby zastosowania tych metod do identyfikacji paciorkowców próchnicotwórczych (6, 7, 8).
Celem badań było porównanie dwóch metod identyfikacji paciorkowców próchnicotwórczych: metody reakcji łańcuchowej polimerazy (ang. polymerase chain reaction, PCR) oraz metody opartej na określaniu biochemicznych właściwości tych bakterii z zastosowaniem testów Rapid ID 32 Strep.
MATERIAŁ I METODY
Materiał do badań stanowiło 495 próbek śliny stymulowanej dzieci w wieku 12 lat, uczęszczających do różnych szkół podstawowych na terenie Polski. Do wyhodowania kolonii bakterii z grupy S. mutans wykorzystano zestaw Dentocult SM Strip mutans (Orion Diagnostica, Finlandia). Następnie, uzyskane kolonie zostały poddane identyfikacji za pomocą zestawu Rapid ID 32 Strep (bioMerieux, Francja) i metodą PCR (9).
W tym celu wyrosłe kolonie bakteryjne przesiewano z podłoża transportowo-hodowlanego Dentocult SM na podłoże Columbia agar z dodatkiem 5% krwi baraniej oraz inkubowano w atmosferze 5% CO 2 w temperaturze 37°C przez 48 godzin. Wszystkie kolonie, które na podstawie preparatu i morfologii na podłożu krwawym podejrzewano o przynależność do rodzaju Streptococcus identyfikowano za pomocą testu Rapid ID 32 Strep zgodnie z instrukcją producenta.Testy Rapid ID 32 Strep umożliwiają identyfikację paciorkowców i gatunków pokrewnych na podstawie ich cech biochemicznych, przy zastosowaniu swoistych testów enzymatycznych (ryc. 1 – str. 16). Pasek testowy Rapid ID 32 Strep składa się z 32 zagłębień zawierających suche substraty, których wyniki po 4 godzinach hodowli z zawiesiną bakterii można odczytać w automacie ATB Expression lub wizualnie.
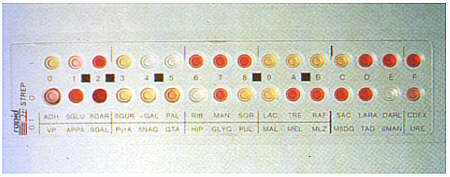
Ryc. 1. Identyfikacja szczepów z rodzaju Streptococcus przy użyciu testu RAPID ID 32 STREPT.
Równolegle, bezpośrednio na materiale wyhodowanym przy pomocy zestawu Dentocult, przeprowadzono reakcję łańcuchową polimerazy PCR ze starterami specyficznymi dla gatunku Streptococcus mutans i Streptococcus sobrinus. Kolonie wyhodowane na podłożu Dentocult zawieszano w sterylnej wodzie i poddawano sonikowaniu, w wyniku którego następowało otwarcie komórek bakteryjnych za pomocą ultradźwięków i uwolnienie DNA. Powstała mieszanina była podstawą reakcji łańcuchowej polimerazy (PCR). Do identyfikacji szczepów S. mutans i S. sobrinus wykorzystano startery opracowane przez Oho i wsp. (9). Po zakończeniu reakcji PCR otrzymane produkty poddawano elektroforezie w żelu agarozowym, a następnie barwiono bromkiem etydyny, który łącząc się z DNA umożliwia jego obserwację w świetle ultrafioletowym (ryc. 2). W przypadku obecności w mieszaninie reakcyjnej DNA szczepów S. mutans otrzymywano na żelu produkt wielkości 517 par zasad, powstały przy zastosowaniu starterów specyficznych do genu gtf B. W obecności DNA pochodzącego od S.sobrinus powstawał produkt o wielkości 712 par zasad, komplementarny do genu gtf I.
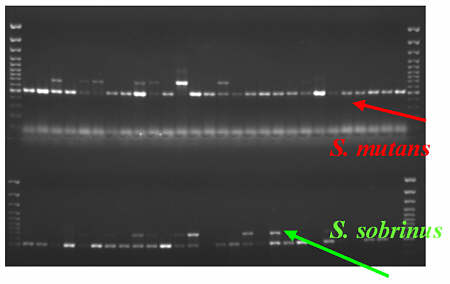
Ryc. 2. Produkty reakcji PCR ze starterami specyficznymi dla S. mutans i S. sobrinus.
WYNIKI
Powyżej zamieściliśmy fragment artykułu, do którego możesz uzyskać pełny dostęp.
Mam kod dostępu
- Aby uzyskać płatny dostęp do pełnej treści powyższego artykułu albo wszystkich artykułów (w zależności od wybranej opcji), należy wprowadzić kod.
- Wprowadzając kod, akceptują Państwo treść Regulaminu oraz potwierdzają zapoznanie się z nim.
- Aby kupić kod proszę skorzystać z jednej z poniższych opcji.
Opcja #1
29 zł
Wybieram
- dostęp do tego artykułu
- dostęp na 7 dni
uzyskany kod musi być wprowadzony na stronie artykułu, do którego został wykupiony
Opcja #2
69 zł
Wybieram
- dostęp do tego i pozostałych ponad 7000 artykułów
- dostęp na 30 dni
- najpopularniejsza opcja
Opcja #3
129 zł
Wybieram
- dostęp do tego i pozostałych ponad 7000 artykułów
- dostęp na 90 dni
- oszczędzasz 78 zł
Piśmiennictwo
1. Loesche W.J.: Role of Streptococcus mutans in human dental decay. Microbiol Revy, 1986, 50, 353-380. 2. Bratthall D., Köhler B.: Streptococcus mutans serotypes: some aspects of their identification, distribution antigenic shifts, and relation to caries. J. Dent. Res., 1976, 55, 15-21. 3. Fujiwara T. et al.: Caries prevalence and salivary mutans streptococci in 0-2 year children in Japan. Community Dent. Oral Epidemiol. 1991, 19, 151-154. 4. Hirose H. et all: Close association between Streptococcus sobrinus in saliva of young children and smooth surface caries increment. Caries Res., 1993, 27, 292-297. 5. Humble M.W. et al.: Api ZYM: a simple rapid system for the detection of bacterial enzymes. J. Clin. Path., 1977, 30, 275. 6. Fordymacki P. i wsp.: Ocena przydatności wybranych systemów komercyjnych do identyfikowania ziarenkowców z rodzaju Streptococcus. Med. Dośw. Mikrobiol., 1988, 50, 171-177. 7. Russel RR.: The application of molecular genetics to the microbiology of dental caries. Caries Res., 1994, 28, 269-282. 8. Igrashi T. et al.: Direct detection of S. m. in human dental plaque by polymerase chain reaction. Oral. Microbiol. Immunol., 1996, 11, 5, 294. 9. Oho T. et all.: Simple and rapid detection of S. m. and S. sobr. in human saliva by PCR. Oral Microbiol. Immunol., 2000, 15, 258-262. 10. Gold O.G. et al.: A selective medium for Streptococcus mutans. Arch.Oral Biol., 1973,18, 1357-1364. 11. Beighton D. at al: A simple biochemical scheme for the differentiation of S. m. and S. sobrinus. Caries Res., 1991,25, 174-178. 12. de Soet J.J.P. et al.: Enumeration of mutans streptococci in clinical samples by using monoclonal antibodies. J. Clin. Microbiol., 1990, 28, 2467-2472. 13. Hamada S., Slade HD.: Biology immunology and cariogenicity of Streptococcus mutans. Microbiol. Rev., 1980, 44, 331-384. 14. Smorawińska M., Kuramitsu HK.: DNA probes for detection of cariogenic Streptococcus mutans. Oral Microbiol. Immunol., 1992, 7, 177-181. 15. Cangelosi GA. et all.: Oligonucleotide probes for mutans streptococci. Mol. Cell Probes, 1994, 8, 73-80. 16. Igrashi T. et al.: Rapid identyfication of mutans streptococcal species. Microbiol. Immunol., 1996, 40,11, 867.


