© Borgis - Postępy Nauk Medycznych 7-8/2007, s. 282-286
*Hanna Makuch-Łasica, Grażyna Nowak, Mirosław Majewski, Krzysztof Warzocha
Częstość krzyżowej rearanżacji genów kodujących receptor T-komórkowy u chorych na ostrą białaczkę limfoblastyczną B-komórkową*
The frequency of cross-lineage T-cell receptor gene rearrangements in B-cell acute lymphoblastic leukemia
Klinika Hematologii Instytutu Hematologii i Transfuzjologii w Warszawie
Kierownik Kliniki: prof. dr hab. n. med. Krzysztof Warzocha
Streszczenie
Wstęp. Rearanżacje genów kodujących części zmienne łańcuchów receptora limfocytów T (TCR) oraz immunoglobulin (IG) mogą być swoistymi markerami w monitorowaniu leczenia ostrych białaczek limfoblastycznych (OBL). W pracy określono częstość klonalnej rearanżacji genów IG i TCR u dorosłych chorych na OBL wywodzącej się z prekursorów limfocytów B (OBL-B) (tzw. krzyżowa rearanżacja).
Materiały i metody. U 36 dorosłych chorych na OBL-B wykonano badania molekularne RT-PCR na obecność genów fuzyjnych, w tym BCR-ABL, TEL-AML i MLL-AF4. Badanie rearanżacji genów łańcuchów ciężkich (IGH) i lekkich kappa/lambda (IGK/IGL) immunoglobulin (IG) oraz receptora T-komórkowego (TCR) gamma (TCRG), TCR delta (TCRD) i TCR beta (TCRB) przeprowadzono metodą multiplex PCR i analizą heterodupleksową. Startery i warunki reakcji dla oceny genów fuzyjnych opracowano zgodnie z protokołem BIOMED-1, a dla rearanżacji genów IG/TCR według protokołu BIOMED-2.
Wyniki. Obecność genów fuzyjnych stwierdzono u 36% chorych z OBL-B, w tym BCR-ABL u 25%, TEL-AML1 u 8% i MLL-AF4 u 3%. Klonalne rearanżacje genów kodujących IG wykryto we wszystkich badanych przypadkach. Części zmienne TCR wykryto u 97% chorych, w tym TCRG i TCRD u 58% oraz TCRB u 47% chorych. Istotnym było wykrycie monoklonalnych rearanżacji TCR u chorych, u których nie wykryto obecności genów fuzyjnych.
Wnioski. Badanie rearanżacji genów TCRB, TCRG i TCRD może służyć jako marker w wykrywaniu i monitorowaniu choroby resztkowej (MRD) u większości dorosłych chorych na OBL-B, szczególne w przypadkach, w których nie wykryto innych aberracji cytogenetycznych.
Summary
Background. T-cell (TCR) and immunoglobulin (IG) variable gene rearrangements are patient-specific targets for clonality studies and monitoring of acute lymphoblastic leukemia (ALL). The aim of our study was to investigate the incidence of clonal TCR and IG genes rearrangements in adult precursor B-cell ALL (B-ALL) (cross-lineage rearrangements).
Materials and methods. The study included 36 newly diagnosed B-ALL patient studied by RT-PCR for the presence of fusion genes, including BCR-ABL, TEL-AML i MLL-AF4 as well as cross-lineage rearrangements of T-cell receptor (TCR) and immunoglobulin (IG) genes. Rearrangements of heavy (IGH) and light kappa/lambda (IGK/IGL) as well as TCR beta (TCRB), TCR gamma (TCRG), and TCR delta (TCRD) genes were analyzed by multiplex PCR in combination with heteroduplex analysis in polyacrylamide gel. Primers and PCR reactions of fusion genes were performed according to BIOMED-1 protocol and rearrangements of IG/TCR genes using BIOMED-2 protocol.
Results. Fusion genes were found in 36% of ALL-B, patients including 25% BCR-ABL, 8% TEL-AML1, and 3% MLL-AF4. Clonal rearrangements of IG genes were found in all studied patients, while cross-lineage TCR genes rearrangements were detected in 97% of B-ALL patients, including 58% TCRG and TCRD genes and 47% TCRB. It is worth noting that cross-lineage TCR gene rearrangements were found in patients with no fusion genes.
Conclusions. The heteroduplex PCR data indicate that TCRB, TCRG and TCRD are suitable targets for minimal residual disease (MRD) monitoring in the majority of adult B-ALL patients. This method seems especially recomended for patients with no other clonal abnormalities.

Wstęp
Ostre białaczki limfoblastyczne (OBL) powstają w wyniku klonalnego rozrostu nowotworowego prekursorów limfocytów i stanowią około 20% wszystkich ostrych białaczek u dorosłych. W grupie OBL, białaczki limfoblastyczne B-komórkowe (OBL-B) stanowią 75-80%, a T-komórkowe 20% (1, 2, 3). Komórki białaczkowe OBL cechuje obecność charakterystycznych markerów różnicowania w zależności od stopnia ich dojrzałości (2). W rozpoznawaniu i monitorowaniu leczenia OBL wykorzystywane są metody morfologiczne, cytochemiczne, immunofenotypowe, cytogenetyczne oraz molekularne (1, 3).
Wykrycie w czasie rozpoznania choroby aberracji chromosomalnych i powstałych w ich wyniku genów fuzyjnych pozwala wykorzystać je jako marker do monitorowania skutków leczenia. Ujawnienie na podstawie tych zmian choroby resztkowej (MRD) pozwala zakwalifikować chorych do grupy o niskim, pośrednim lub wysokim ryzyku wznowy (2). Metody molekularne stosowane w monitorowaniu leczenia cechują się większą czułością (10-4-10-6) niż stosowane powszechnie metody cytomorfologiczne (10-2-10-3) i cytometrii przepływowej (10-3-10-4) (4).
W rozpoznawaniu i monitorowaniu wyników leczenia metodami molekularnymi chorób limfoproliferacyjnych użyteczne jest również badanie klonalnej rearanżacji genów kodujących części zmienne immunoglobulin (IG) oraz receptora T-komórkowego (TCR) (1, 4-14). W trakcie dojrzewania limfocytów B dochodzi do rearanżacji genów segmentowych V (variable), D (diversity) i J (joining) kodujących części zmienne łańcuchów ciężkich (IGH) i lekkich kappa (IGK), lambda (IGL) immunoglobulin. Proces rearanżacji polega na delecji fragmentów DNA i utworzeniu struktury V(D)J, kodującej część zmienną łańcuchów immunoglobulin (9).
W OBL-B dochodzi również do rearanżacji genów zmiennych V(D)J kodujących łańcuchy receptora limfocytów T - TCR (krzyżowa rearanżacja) (5-8). Komórki klonu białaczkowego mają w identyczny sposób zrearanżowane geny IG/TCR. Geny V(D)J TCR rzadko podlegają procesowi somatycznej hypermutacji i cechują się dużą trwałością zmian genetycznych, niezbędną do monitorowania wyników leczenia na poziomie molekularnym. W nawrocie OBL, u 86-100% chorych wykrywa się identyczny rodzaj rearanżacji genów IG/TCR, jak obserwowany na początku choroby (7, 9, 13, 14).
Klonalną rearanżację genów IGH, IGK, TCR delta (TCRD), TCR gamma (TCRG) i TCR beta (TCRB) wykrywa się odpowiednio u 98%, 50%, 40-46%, 55-58% i 27% chorych dzieci na OBL-B. Dodatkowo stwierdza się u ponad 90% chorych obecność przynajmniej dwu rodzajów rearanżacji, które mogą być użyte do wykrywania i monitorowania choroby resztkowej. Wykazano również związek między częstością występowania niektórych rodzajów rearanżacji, a wiekiem chorego i obecnością specyficznych aberracji chromosomowych (4, 7, 15).
Badanie rearanżacji genów IG/TCR może być wykonywane metodą Southern blot, uważaną do niedawna za złoty standard lub techniką multipleks PCR. Metoda Southern blot jest czasochłonna, wymaga drogiego wyposażenia oraz dużej ilości i dobrej jakości DNA. Metoda multipleks PCR jest szybka, wymaga niewielkiej ilości DNA, które można uzyskać nawet z materiału utrwalonego i zatopionego w parafinie (7, 9,16). Czułość metody multipleks PCR, w zależności od rodzaju rearanżacji, waha się od 5 x 105 do 10-5 (1, 4, 9, 17, 18).
Przedmiotem obecnych badań była ocena częstości rearanżacji genów kodujących części zmienne łańcuchów TCR dla wykazania, czy parametr ten mógłby spełniać kryteria odpowiednie dla monitorowania MRD u dorosłych chorych na OBL-B.
Materiał i metody
Pacjenci. Materiał do badań pochodził od 36 dorosłych chorych z nowo rozpoznaną OBL-B, w tym 19 kobiet i 17 mężczyzn (wiek 18-87 lat, mediana 39,5 lat). Rozpoznanie ustalono na podstawie badań cytomorfologicznych, cytochemicznych i immunofenotypowych (3).
Badania genów fuzyjnych i rearanżacji genów IG/TCR. U wszystkich 36 chorych z OBL-B wykonano badania molekularne na obecność genów fuzyjnych, w tym BCR-ABL, TEL-AML1 i MLL-AF4 oraz rearanżacji genów IG/TCR, w tym IGH, IGK i IGL oraz TCRD, TCRG i TCRB. Materiał do badań stanowiły próbki krwi obwodowej lub szpiku kostnego, z których izolowano komórki jednojądrzaste na gradiencie gęstości 1,077 g/mL (Gradisol L firmy Biomed). DNA izolowano metodą kolumienkową z wykorzystaniem zestawu firmy Qiagen QIAamp DNA Blood Mini Kit zgodnie z zaleceniami producenta. Startery i warunki reakcji dla oceny genów fuzyjnych opracowano zgodnie z protokołem BIOMED-1, a dla rearanżacji genów IG/TCR według protokołu BIOMED-2 (9,19).
Reakcje PCR prowadzono w termocyklerze SpecGene Techne w końcowej objętości 25 ?l (50-100 ng DNA, ABI Buffer II, 10 pmol każdego ze starterów 3´ i 5´, dNTP w stężeniu końcowym 200 ?mol/L, MgCl2 w stężeniu końcowym 1,5-3,0 mmol/L, polimeraza AmpliTaq Gold 0,5-1 IU na jedną reakcję). Warunki reakcji PCR polegały na wstępnej aktywacji (7 min. w 95°C) i kolejnych 35 cyklach amplifikacji, każdy złożony z fazy denaturacji (30 s w 94°C), przyłączania (30 s w 60°C), wydłużania (30 s w 72°C) oraz końcowego wydłużania (10 min. w 72°C).
W trakcie każdego eksperymentu wykonywano kontrolne badania PCR, w tym dodatnie (DNA izolowane z linii komórkowej Jurkat, DNA pacjentów, w którym wykazano obecność monoklonalnych rearanżacji) i kontrole ujemne (bez DNA oraz DNA dawców bez choroby limfoproliferacyjnej) (9, 16). Identyfikacja klonalnej rearanżacji genów kodujących część zmienną TCRB została wykonana w trzech multipleksowych reakcjach PCR, w tym TCRB-A z użyciem 23 starterów Vβ2-24 i 9 starterów Jβ1.1-2.7, TCRB-B z użyciem 23 starterów Vβ2-24 i 4 starterów Jβ2.1-2.5 i TCRB-C z użyciem 2 starterów Dβ1 i Dβ2 oraz 13 starterów Jβ1.1-2.7 (9). Identyfikacja klonalnej rearanżacji genów kodujących część zmienną łańcucha TCRG została wykonana w dwóch multipleksowych reakcjach PCR, w tym TCRG-A z użyciem dwu starterów VγIf i Vγ10 oraz dwóch Jγ1.1/2.1 i Jγ1.3/2.3 oraz TCRG-B z użyciem dwóch starterów Vγ9 i Vγ11 oraz dwóch Jγ1.1/2.1 i Jγ1.3/2.3 (9). Identyfikacja klonalnej rearanżacji genów kodujących część zmienną łańcucha TCRD została wykonana w jednej multipleksowej reakcji PCR z użyciem 7 starterów Dδ1-6 i Dδ2 oraz 5 starterów Jδ1-4 i Dδ3 (9).
Produkty PCR poddawano analizie heterodupleksowej przez denaturację w 95°C przez 5 min., a następnie ponowną renaturację w 4°C przez 60 min. Po analizie heterodupleksowej produkty PCR rozdzielano w elektroforezie pionowej, w 6% żelu poliakrylamidowym i 0,5 x buforze TBE (Tris-Borate-EDTA) przez 50 min., przy napięciu 150 mV. Żele po rozdziale barwiono 10 min. w roztworze bromku etydyny o stężeniu 0,5 ?g/mL. Wyniki odczytywano w świetle lampy UV (ryc. 1).
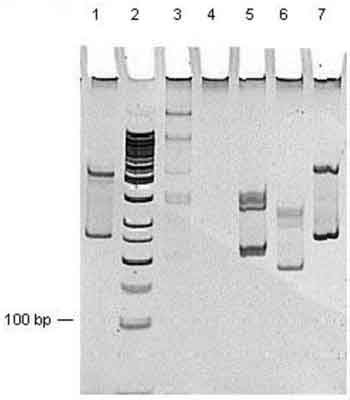
Ryc. 1. Wynik elektroforezy w żelu poliakrylamidowym z rozdziałem produktów multipleks PCR i analizy heterodupleksowej z badania rearanżacji genów kodujących części zmienne łacuchów TCR u chorego z rozpoznaniem OBL-B. 1 – rearanżacja TCRB-A wynik dodatni, produkt 240-285 bp; 2 – wzorzec 50 bp; 3 – rearanżacja TCRB-B wynik ujemny; 4 – rearanżacja TCRB-C wynik ujemny; 5 – rearanżacja TCRG-A wynik dodatni, produkt 145-255 bp; 6 – rearanżacja TCRG-B wynik dodatni, produkt 80-220 bp; 7 – rearanżacja TCRG-B wynik dodatni, produkt 120-280 bp.
Wyniki
Obecność genów fuzyjnych stwierdzono u 13 z 36 badanych chorych (36%), w tym BCR-ABL u 9 chorych (25%), TEL-AML1 u 3 chorych (8%) i MLL-AF4 u 1 pacjenta (3%).
Klonalne rearanżacje genów kodujących IG wykryto we wszystkich badanych przypadkach, w tym IGH u 33 chorych (92%), IGK u 23 chorych (64%) i IGL u 8 pacjentów (22%).
Powyżej zamieściliśmy fragment artykułu, do którego możesz uzyskać pełny dostęp.
Mam kod dostępu
- Aby uzyskać płatny dostęp do pełnej treści powyższego artykułu albo wszystkich artykułów (w zależności od wybranej opcji), należy wprowadzić kod.
- Wprowadzając kod, akceptują Państwo treść Regulaminu oraz potwierdzają zapoznanie się z nim.
- Aby kupić kod proszę skorzystać z jednej z poniższych opcji.
Opcja #1
29 zł
Wybieram
- dostęp do tego artykułu
- dostęp na 7 dni
uzyskany kod musi być wprowadzony na stronie artykułu, do którego został wykupiony
Opcja #2
69 zł
Wybieram
- dostęp do tego i pozostałych ponad 7000 artykułów
- dostęp na 30 dni
- najpopularniejsza opcja
Opcja #3
129 zł
Wybieram
- dostęp do tego i pozostałych ponad 7000 artykułów
- dostęp na 90 dni
- oszczędzasz 78 zł
Piśmiennictwo
1. Hoelzer D., et al.: Acute Lymphoblastic Leukemia. Hematology (Am. Soc. Hemat. Educ. Program), 2002: 162-192.
2. Copelan E.A., McGuire E.: The Biology and Treatment of Acute Lymphoblastic Leukemia in Adults. Blood 1995; 85: 1151-1168.
3. Devine S.M., Larson R.A.: Acute leukemia in adults: recent developments in diagnosis and treatment. Cancer J. Clin., 1994; 44: 326-352.
4. Brumpt C., et al.: The incidence of clonal T-cell receptor rearrangements in B-cell prekursor acute lymphoblastic leukemia varies with age and genotype. Blood 2000; 96: 2254-2261.
5. Szczepański T., et al.: Cross-lineage T cell receptor gene rearrangements occur in more than ninety percent of childhood precursor-B acute lymphoblastic leukemias: alternative PCR targets for detection of minimal residual diesese. Leukemia 1999; 13: 196-205.
6. Nosaka T., et al.: Cross-lineage gene rearrangements in human leukemic B-precursor cells occur frequently with V-DJ reangements of IgH genes. Blood 1989; 74: 361-368.
7. van der Velden V.H.J., et al.: Age-related patterns of immunoglubulin and T-cell receptor gene rearrangements in precursor-B-ALL: implications for detection of minimal residual disease. Leukemia 2003; 17: 1834-1844.
8. Szczepański T., et al.: Comparative analysis of Ig and TCR gene rearrangements at diagnosis and at relapse of childhood precursor-B-ALL provides improved strategies for selection of stable PCR targets for monitoring of minimal residual disease. Blood; 2002; 99: 2315-2323.
9. van Dongen J.J.M., et al.: Design and standardization of PCR primers and protocols for detection of clonal immunoglobin and T-cell receptor gene recombinations in suspect lymphoproliferations: Report of the BIOMED-2 Concerted Action BMH4-CT98-3936. Leukemia 2003; 17: 2257-317.
10. Germano G., et al.: Clonality profile in relapsed precursor-B-ALL children by GeneScan and sequencing analyses. Consequences on minimal residual disease monitoring. Leukemia 2003; 17: 1573-1582.
11. Szczepański T., et al.: Immunoglobulin and T cell receptor rearrangement patterns in acute lymphoblastic leukemia are less mature in adults than in children: implications for selection of PCR targets for detection of minimal residual disease. Leukemia 1998; 12: 1081-1088.
12. van der Velden V.H.J., et al.: T cell receptor gamma gene rearrangements as rargets for detection of minimal residual disease in acute lymphoblastic leukemia by real-time qquantitative PCR analysis. Leukemia 2002; 16: 1372-1380.
13. van der Velden V.H.J., et al.: TCRB gene rearrangements in childhood and adult precursor-B-ALL: frequency, applicability as MRD-PCR target, and stability between diagnosis and relapse. Leukemia 2004; 18: 1971-1980.
14. Szczepański T., et al.: Ig heavy chain gene rearrangements in T-cell acute lymphoblastic leukemia exhibit predominant DH6-19 and DH7-27 gene usage, can result in complete V-D-J rearrangements, and rare in T-cell receptor αβ lineage. Blood 1999; 93: 4079-4085.
15. Vidriales M-B., et al.: Minimal residual disease in aldolescent (older than 14 years) and adult acute lymphoblastic leukemias: early immunophenotypic evaluation has high clinical valve. Blood 2003, 10: 4695-4700.
16. Sandberg Y., et al.: BIOMED-2 multiplex immunoglobulin/T-cell receptor polymerase chain reaction protocols can reliably replace Southern blot analysis in routine clonality diagnostics. J. Mol. Diag. 2005; 7: 495-503.
17. Germano G., et al.: Rapid detection of clonality in patients with acute lymphoblastic leukemia. Haematologica 2001; 86: 382-385.
18. Malec M., et al.: Analysis of minimal residual disease in childhood acute lymphoblastic leukemia: comparison between RQ-PCR analisis of Ig/TCR gene rearrangements and multicolor flow cytometric immunophenotyping. Leukemia 2004; 18: 1630-1636.
19. van Dongen J.J.M., et al.: Standarized RT-PCR analysis of fusion gene transcripts from chromosome aberrations in acute leukemia for detection of minimal residual disease. Report of the BIOMED-1: Investigation of minimal residual disease in acute leukemia. Leukemia 1999; 13: 1901-1928.
20. Evans P.A.S., et al.: Significantly improved PCR-based clonality testing in B-cell malignancies by use of multiple immunoglobulin gene targets. Report of the BIOMED-2 Concerted Action BHM4-CT98-3936. Leukemia 2007; 21: 207-214.
21. Meleshko A.N., et al.: Rearrangements of IGH, TCRD and TCRG genes as clonality marker of childhood acute lymphoblastic leukemia. Exp. Oncol. 2005; 27, 4: 319-324.
22. Sandberg Y., et al.: Leukemia 2007; 21: 230-7.
23. Szczepański T. et al: Vδ2-Jα rearrangements are frequent in precursor-B-acute lymphoblastic leukemia but rare in normal lymphoid cells. Blood 2004; 103: 3798-3804.

